Der genetische Inzuchtkoeffizient
19.07.2022 08:38von Astrid Hübner
Der Inzuchtkoeffizient (IK) gibt die Wahrscheinlichkeit an, dass an einem Lokus beide Allele vom selben Vorfahr stammen.
Er soll die Inzucht eines Individuums messen.
Im englischen wird er als „COI – Coefficient of Inbreeding“ bezeichnet, im französischen als „coefficient de consanguinité“.
Viele Hunde-Datenbanken berechnen den auf der Ahnentafel basierenden IK bereits und deshalb ist er den meisten Haltern und Züchtern ein Begriff. Im Beitrag Inzuchtkoeffizient (IK) und Ahnenverlustkoeffizient (AVK) erfährst du mehr über die herkömmliche Berechnung
Je höher die Inzucht desto geringer die Diversität eines Hundes.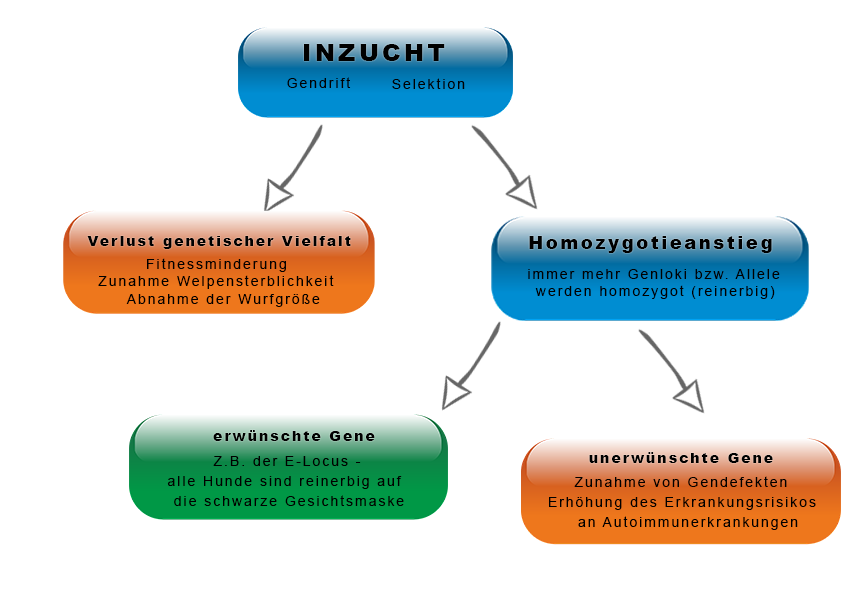
In der Populationsgenetik hat Fitness nichts mit der Sportlichkeit zu tun. „Survival oft he fittest“ (Darwin) bedeutet „das Überleben des Best-angepassten“
Darwin (1860) beschäftigte sich Mitte des 19. Jahrhunderts mit dem Thema und erkannte damals schon die Chancen und vor allem Gefahren der Inzucht:
Zitat
„…dass enge Inzucht zwischen den nächsten Verwandten einige Generationen lang fortgesetzt, zumal wenn dieselben unter
gleichen Lebensbedingungen gehalten werden, endlich schwache und unfruchtbare Sprosslinge liefert.“
Wir haben die Wurfgrößen der einzelnen Varietäten mit dem durchschnittlichen genetischen Inzuchtkoeffizienten (gIK oder gCOI) verglichen. Hier sieht man deutlich, dass die Wurfgrößen mit steigendem gIK abnehmen. Für diese Statistik haben wir die durchschnittlichen Wurfgrößen der SCC - 8324 Würfe 2001-2019 - genommen (blauer Balken) und die Wurfgrößen eines deutschen Zuchtverbandes - 252 Würfe 2009 – 2021 (grüner Balken).
Die gelbe Linien gibt den durchschnittlichen gIK an, den das Labor Embark als Durchschnitt für die jeweilige Varietät angibt.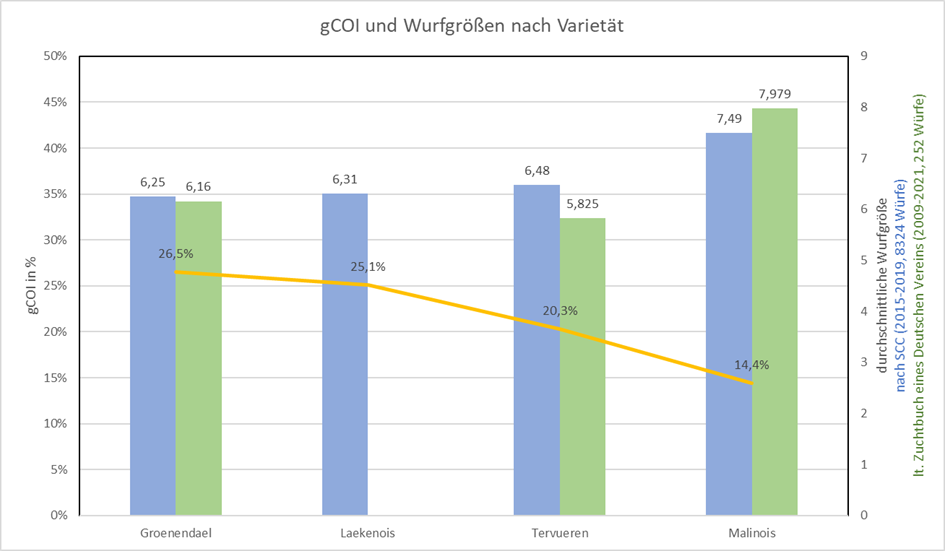
Die Statistik zeigt, dass auch beim Belgier mit steigender Inzucht die Wurfgrößen kleiner werden.
Die genetische Diversität bzw. genetische Vielfalt aufrecht zu erhalten sollte also das oberste Ziel in der Zucht sein.
Die herkömmliche Berechnung des IK basiert auf der Ahnentafel eines Hundes. Im Idealfall ist der Stammbaum bis zur Gründung der Rasse vollständig. In Wirklichkeit gehen die meisten Ahnentafeln jedoch nur auf 5 bis 10 Generationen zurück und gerade beim Malinois stimmen die Ahnentafeln oft nicht. Die meisten IK-Rechner gehen davon aus, dass die ursprünglichen Vorfahren im Stammbaum keine Beziehung zueinander haben. Daher kann ein Ahnentafel-IK auf 5 Generationen berechnet viel niedriger sein als der IK, der auf 10 Generationen berechnet wurde. Und dieser ist wahrscheinlich viel niedriger als der wahre IK, wenn der vollständige Stammbaum bis zu den Rassengründer bekannt ist. Aus diesem Grund gibt es keine Antwort darauf, was ein „guter“ IK ist. Es hängt alles davon ab, wie vollständig die Ahnentafel ist.
Das nächste Problem ist, dass nach herkömmlicher Berechnung alle Wurfgeschwister den gleichen IK haben, obwohl sie sehr wahrscheinlich unterschiedliche Gene geerbt haben.
Aufgrund des Segregationsprinzips können zwei Hunde mit identischen erwarteten IKs aus einem Stammbaum sehr unterschiedliche Inzuchtniveaus aufweisen. Dies hängt davon ab, welche Individuen welche Chromosomensegmente erben.
Mittlerweile sind Labore in der Lage, einen genetische Inzuchtkoeffizienten zu berechnen.
Der genetische Inzuchtkoeffizient ist die genaueste Methode zur Messung der Inzucht. Im Gegensatz zu Stammbaum-basierten IK-Berechnungen bewertet der genetische IK die tatsächlichen DNA-Stücke des Hundes, um festzustellen, welcher Anteil auf Inzucht zurückzuführen ist. Der genetische IK kann Inzucht in weit mehr vergangenen Generationen erkennen sowie bessere Informationen liefern, als dies über Stammbaumberechnungen möglich ist.
Diese Messung erfolgt über Marker den SNPs (Snips gesprochen) „Single Nucleotide Polymorphism“.Wie funktioniert das?
Die gebräuchlichste Methode zur Bestimmung von Inzucht anhand von DNA-Daten verwendet eine Technik, die die Menge an Inzucht quantifiziert, die in „Läufen von Homozygotie“ (ROH) dargestellt wird, das sind Regionen der Chromosomen, in denen es viele aufeinanderfolgende homozygote Loci gibt ( Ceballos ua 2018; Curik ua 2014).
Die durch Inzucht erzeugte Homozygotie ist nicht zufällig über die Chromosomen verstreut. Sowohl die natürliche als auch die künstliche Selektion wird „Hot Spots“ der Homozygotie in den Regionen des Chromosoms erzeugen, in denen Gene selektiert werden (Sams & Boyko 2018).
Embark stellen ihre Marker graphisch (stark vereinfacht) dar. Für uns ist wichtig zu verstehen, dass es sich nie um das gesamte Chromosom handelt in den Abbildungen, sondern NUR um die Marker, die sich darauf befinden.
Alle Inzuchtbereiche werden farbig in gelb angezeigt. Die grauen Bereiche sind mischerbig. Inzuchtbereiche bedeuten, dass der Hund von beiden Elternteilen an dieser Stelle in seinem Genom dasselbe DNA-Stück (identisch durch Abstammung) geerbt hat. Je mehr Inzucht ein Hund hat, desto bunter erscheint dieses Diagramm.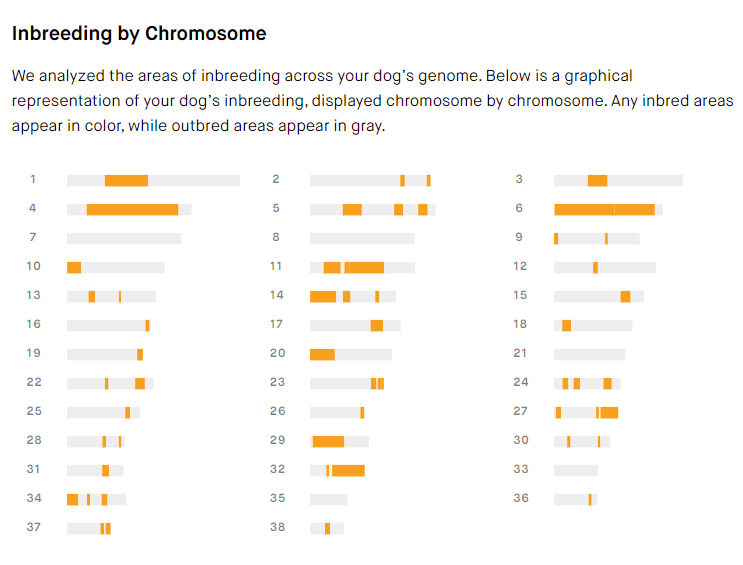
Hündin: Genetischer IK 23% / 2 -3 Inzucht
Da man weiß, dass sich die Blöcke der Homozygotie als Ergebnis von Inzucht bilden, kann man sie verwenden, um den Anteil der Inzucht im gesamten Genom abzuschätzen.
Neue vs. ältere Inzucht
Man kann anhand von diesen Daten sogar erkennen, ob es sich um neue oder alte Inzucht handelt. Man weiß, dass Inzucht immer längere Serien von Homozygotie produziert. Aber in jeder Generation kommt es während der Meiose zu einem Crossover, bei dem Abschnitte von Chromosomen ausgetauscht werden. Wenn in einem Homozygotieblock Brüche auftreten, wird ein größerer Homozygotielauf in zwei kleinere aufgeteilt. Daher ist zu erwarten, dass die Blöcke im Laufe der Generationen kürzer werden, d.h. die älteste Inzucht ist durch die kürzesten Blöcke belegt.
Obwohl allgemein Einigkeit darüber zu herrschen scheint, dass ROH derzeit die Methode der Wahl zur Schätzung des Inzuchtniveaus aus DNA ist, gibt es immer noch Diskussionen über die Größe der Blöcke der Homozygotie, die in die Berechnung einzubeziehen sind (Sams & Boyko 2018). Sicherlich stellen die längsten Blöcke die jüngste Inzucht dar und sollten berücksichtigt werden, aber sollte auch ältere Inzucht einbezogen werden? Dies kann zu einem erheblichen Unterschied in den Inzuchtschätzungen führen, und bis jetzt gibt es keinen Konsens darüber, was die beste Schätzung liefert.Unterschiede zwischen Wurfgeschwistern
Diese Hunde stammen alle aus einem Wurf: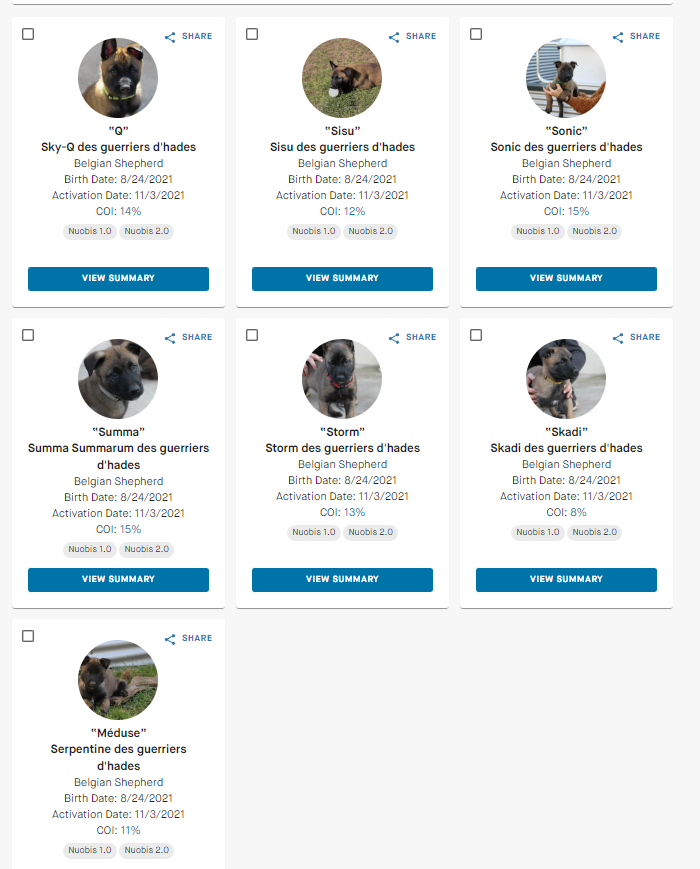
Die Werte für den gCOI liegen bei den Geschwistern zwischen 8 und 15%
Um nochmal zu verdeutlichen, wie unterschiedlich zwei Hunde aus einem Wurf ausfallen können, haben wir zwei Auswertungen übereinander gelegt.
Die eine Hündin hat einen GIK von 10%, die andere von 12%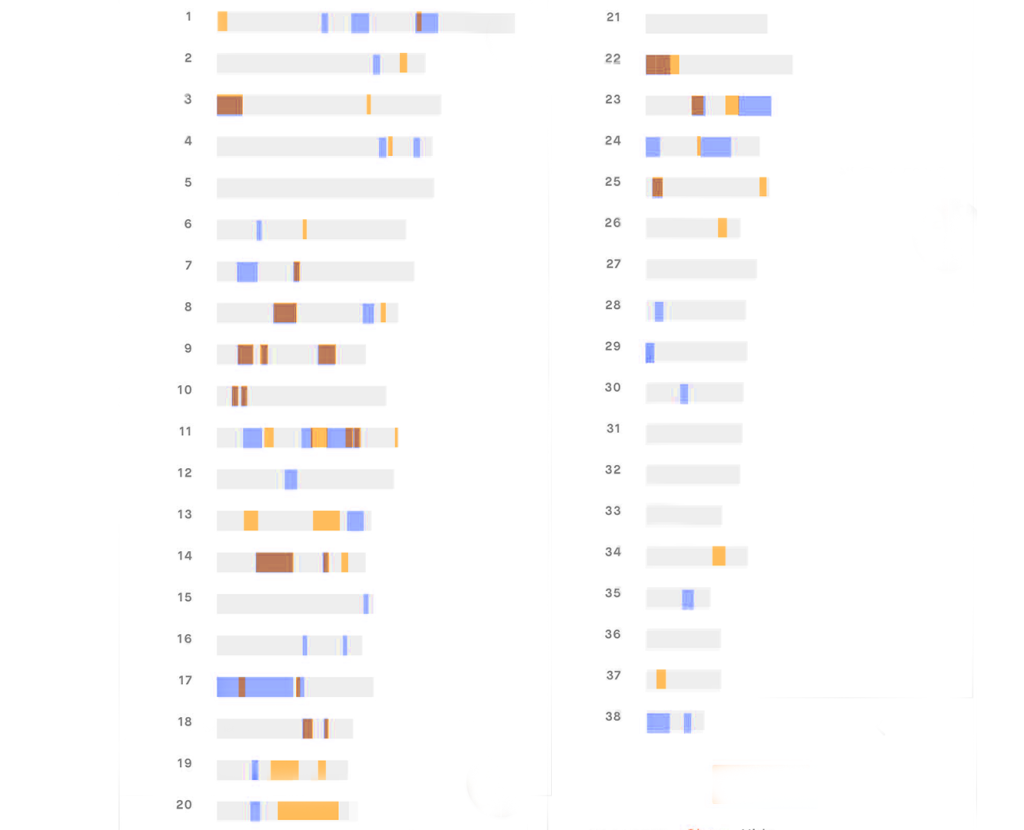
Blau Hündin 1 – gelb Hündin 2 – braun Überschneidungen der beiden Hündinnen
Diese Gegenüberstellung verdeutlicht nochmal, dass die herkömmliche Errechnung des IK über die Ahnentafel sehr wenig aussagt. Nur in den braunen Teilen sind die Hündinnen beide homozygot.Wie kann ich erfahren, wie hoch der gIK meines Hundes ist?
Im Moment gibt es zwei Labore, die den gIK auswerten. Feragen berechnet den gIK auf 6 Generationen, Embark „genomweit“, schließt also ältere Inzucht mit ein. Wie bereits erwähnt gibt es derzeit kein „besser oder schlechter“, wie weit ausgewertet wird. Es bedeutet aber, dass die Werte nicht miteinander verglichen werden können.
Beispiele Feragen:
Beispiele Embark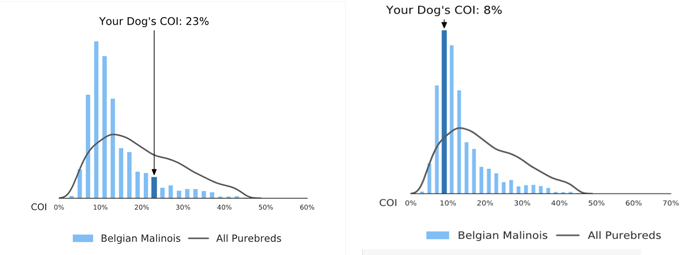
Wie sieht es bei der gesamten Population aus?
In seiner Studie von 2016 liegen Groenendael, Tervueren und Malinois auf und über dem genetischen Inzuchtniveau von Vollgeschwister-verpaarungen
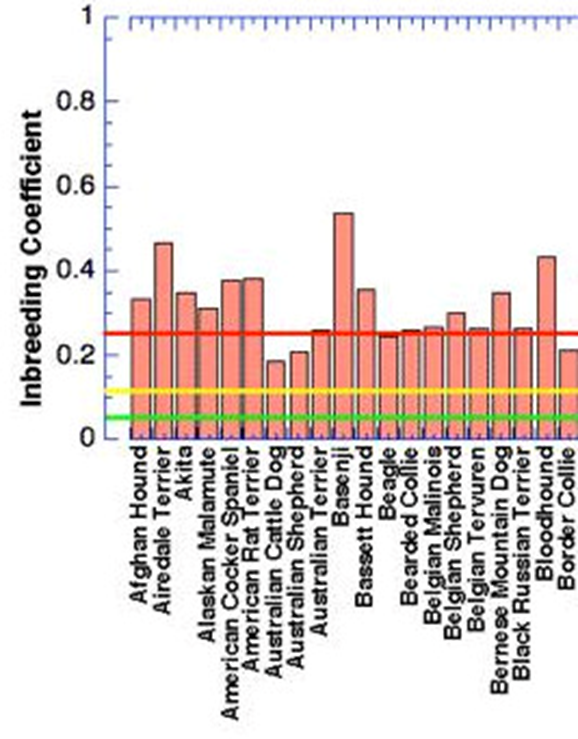
Quelle: Dayna Dreger out of Elaine Ostrander's lab at NIH (2016)
Die rote Linie entspricht einer Vollgeschwisterverpaarung (25 %)
Die gelbe Linie entspricht einer Halbgeschwisterverpaarung. (12,5 %)
Die grüne Linie entspricht einer Verpaarung von Cousin und Cousine (6,25 %)
Die veröffentlichten Daten von Embark kann man nicht als Wert für eine geschlossene Population nehmen. In die Berechnung von ihnen fließen nämlich alle Belgier ein, die genetisch der Rasse Malinois zugeordnet werden, unabhängig davon, ob die Hunde aus einem geschlossenen Zuchtbuch kommen. Auch werden Intervarietäten als Mischlinge behandelt.
Die Datenmenge von Feragen ist derzeit noch zu gering, um einen repräsentablen Durchschnitt zu errechnen.
Das Belgian Shepherd Health project hat aus knapp 600 Belgiern folgende Durchschnittswerte (Embark) errechnet:
Schaulinien 26,8 %, Kombi 17,4 %, Arbeitlinien 12,6 %
Der höchste gIK beim Groenendael betrug 67 %, beim Tervueren 40 %, beim Malinois 37 % und beim Laekenois 21 %
Der niedrigste gIK, der gefunden wurde war 4% (Intervarietät/Arbeitslinie)
Was sind die Gründe für die teilweise sehr hohe Inzucht?
Einer der wichtigsten Faktoren ist die Größe der Tierpopulation. Große Populationen verändern sich langsamer und sind im Laufe der Zeit vorhersehbarer; kleine Populationen sind genetisch instabil, weil sie empfindlicher auf Zufallseffekte reagieren und weil Veränderungen sehr schnell eintreten können.
Die vier belgische Varietäten unterscheiden sich in der Populationsgröße erheblich.
Innerhalb der FCI gelten vier Varietäten als eine Rasse und eine Kreuzung ist mit Genehmigung zulässig. In den USA werden sie als separate, "reine" Rassen behandelt.
Ob wir sie als getrennte Rassen oder nur als Varietäten behandeln, hat tiefgreifende Auswirkungen auf die Genetik der Populationen. In kleinen Populationen steigt die Inzucht schneller an. Dies ist vorhersehbar und unvermeidlich. Zusammenfassend kann man festhalten, dass die Kreuzung der Varietäten dazu beiträgt, die Populationsgröße zu erhöhen, welche langfristig dafür verantwortlich ist, wie stark der Inzuchtgrad der Rasse steigt.
Bei einer Populationsgröße von 100 steigt nach 50 Generationen der IK auf 23%
Bei einer Populationsgröße von 50 steigt nach 50 Generationen der IK auf 38%
Wir reden hier von einem Prozeß, der OHNE UNSER ZUTUN entsteht, wenn die Tiere rein zufällig verpaart werden. In der Realität verpaaren sie sich aber nicht zufällig. Wir machen gewollte Inzuchten, nutzen immer wieder die gleichen Top-Deckrüden, selektieren stark – ungeachtet ob auf Leistung oder Standard Konformität.
Den Beweis dafür finden Sie in der folgenden Grafik. Die grüne Linie ist das Inzuchtniveau von Generation zu Generation, das vorhergesagt wird, wenn die Tiere zufällig gezüchtet werden. (Es ist keine gerade Linie, weil es die tatsächliche Anzahl von Hunden und ihre Inzucht verwendet, um die nächste Generation vorherzusagen, und nicht eine simulierte Population.)
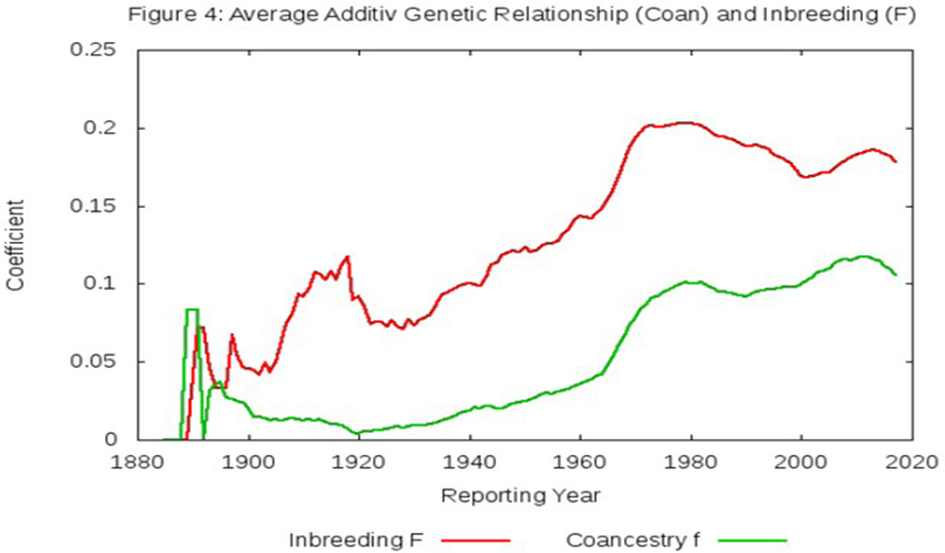
Quelle
Die grüne Linie ist das vorausberechnete Inzuchtniveau von Generation zu Generation, für zufällig verpaarte Tiere. Die rote Linie zeigt die tatsächlich in der Population beobachtete Inzucht.
Die durchschnittliche Inzucht der Population liegt IMMER viel höher als die vorhergesagt für zufällig verpaarte Tiere
Im Laufe des letzten Jahrhunderts hätten Züchter die Inzuchtrate deutlich besser kontrollieren und positiver beeinflussen können, wählten aber stattdessen sie Zuchtpaare aus, die enger als der Durchschnitt miteinander verwandt waren. Ebenso verringert der unnötige Zuchtausschluss grundsätzlich zuchtgeeigneter Tiere die Population. Weitere Faktoren sind der Gründer-Effekt, Flaschenhals Ereignisse und das Popular sire Syndrome.
Mehr dazu könnt ihr in unseren Beiträgen „Effektive Populationsgröße“ und „Die genetische Diversität“ nachlesen.
Welche Möglichkeiten gibt es den gIK meiner Nachzucht zu beeinflussen?
Feragen sowie Embark stellen ein „Matching“ für Zuchtpaare zur Verfügung. Mehr dazu in unserem Beitrag „Matchmaker“ – wie uns die Molekulargentik bei der Zuchtplanung unterstützen kann“ (Artikel in Arbeit)


Quellen:
https://feragen.at/genetische-vielfalt/g...he-diversitaet/ (abgerufen August 2022)
https://my.embarkvet.com/members (abgerufen August 2022)
https://www.biologie-seite.de/Biologie/Inzuchtkoeffizient (abgerufen August 2022)
https://belgianshepherdhealthproject.org/ (abgerufen August 2022)
https://www.mechelaar.de/zucht/ (abgerufen August 2022)
https://www.centrale-canine.fr/lofselect (abgerufen August 2022)
https://pubmed.ncbi.nlm.nih.gov/30429214/ (abgerufen August 2022)
https://www.biologie-seite.de/Biologie/S...en%20bezeichnet. (abgerufen August 2022)
https://www.instituteofcaninebiology.org...-inbreeding-roh (abgerufen August 2022)
Informationen zu diesem Artikel
-
Erstellt von:
Breed a healthy race
Kategorie: Genetik
19.07.2022 08:38:00 Uhr
zuletzt bearbeitet: 15.12.2023 12:06 - Keine Kommentare
Kommentare
Der Captcha wurde falsch eingeben.
Sie haben zu viele Links in Ihrem Beitrag. Maximal dürfen Links gesetzt werden.
Sie haben zu viele Bilder in Ihrem Beitrag. Maximal dürfen Bilder verwendet werden.
Sie haben zu viele animierte Bilder in Ihrem Beitrag. Maximal dürfen animierte Bilder verwendet werden.
Ein in Ihrem Beitrag verwendetes Bild überschreitet die zulässige Breite, die vom Administrator des Forums festgelegt wurde. Die maximal erlaubte Breite sind Pixel.
Ein in Ihrem Beitrag verwendetes Bild überschreitet die zulässige Höhe, die vom Administrator des Forums festgelegt wurde. Die maximal erlaubte Höhe sind Pixel.
Der eingegebene Text ist zu lang (maximal 65.500 Zeichen).
Sie dürfen erst in Tagen Links zu externen Webseiten posten.
Sie dürfen erst nach Beiträgen Links zu externen Webseiten posten
Bitte lösen Sie das Captcha, um auch als Gast Links in diesem Forum zu posten.
Gäste können leider keine Links zu externen Seiten posten.
Bitte entfernen Sie folgende Links, um den Beitrag zu speichern:
